La Red Nacional de Laboratorios Españoles de Secuenciación Genómica de SARS-CoV-2 (RELECOV), coordinada por el Centro Nacional de Microbiología (CNM-ISCIII) ha publicado el informe sobre evolución del virus en España entre octubre de 2022 y octubre de 2023, que revela datos de secuenciación genómica; vigilancia de variantes, linajes y sublinajes; análisis de mutaciones, y estudios filogenéticos.
Entre la información que desprende este documento se ha dado a conocer la circulación de cerca de 800 linajes y sublinajes diferentes de ómicron en España, asignados a diferentes clados del SARS-CoV-2. Los clados son grupos filogenéticos que definen la evolución biológica del virus y su comportamiento, algo fundamental para la vigilancia y su aplicación en salud pública. Todos los linajes y sublinajes detectados son descendientes de la variante ómicron, predominante en España a partir de 2022. El informe revela datos de secuenciación genómica, vigilancia de variantes, análisis de mutaciones y estudios filogenéticos, y aporta datos que permiten conocer mejor cómo se transmite el coronavirus.
Además de poder conocer los diferentes linajes y sublinajes de ómicron, así como llevar un seguimiento de su evolución; durante el año de estudio que refleja este informe se han caracterizado más de 47.000 virus SARS-CoV-2, lo que ha permitido conocer su genoma.
Circulación de las variantes
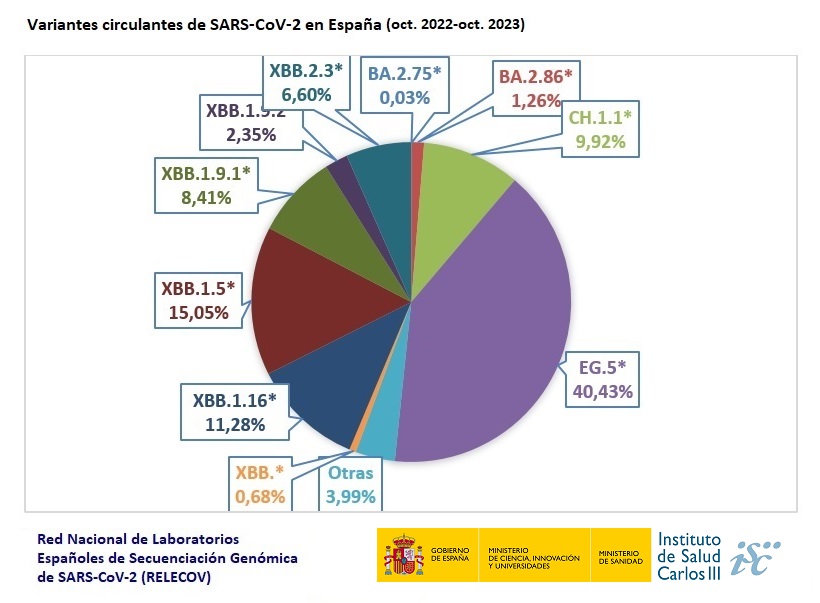
En el periodo estudiado, siguiendo las directrices establecidas por la Organización Mundial de la Salud (OMS), las variantes circulantes de interés (VOI) detectadas pertenecen a los linajes XBB.1.5* (clado 23A), XBB.1.16* (clado 23B) y EG.5* (clado 23F). Por otra parte, las variantes bajo monitorización (VUM) comprenden los linajes correspondientes a BA.2.75* (clado 22D), CH.1.1* (clado 23C), XBB* (clado 22F), XBB.1.9.1* (clado 23D), XBB.1.9.2* (clado 23D), XBB.2.3* (Clado 23E) y BA.2.86*.
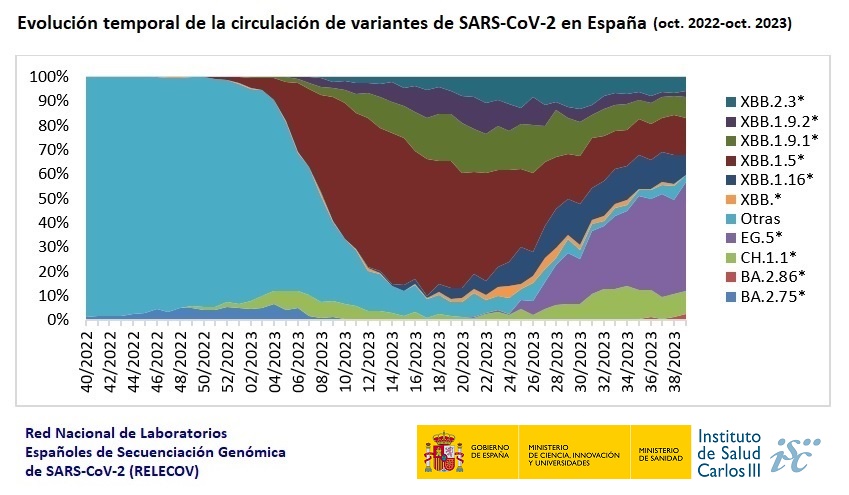
El informe también muestra la evolución de las variantes. Se observó una disminución muy significativa en la circulación de diferentes variantes, entre ellas BA.2.75* (clado 22D), XBB.1.5, XBB.1.9.1 y XBB.1.9.2, y un aumento en la circulación de otras, como es el caso de EG.5, CH.1.16* y CH.1.1*. Del mismo modo, el documento analiza la evolución de la circulación de diversos sublinajes y cómo se han ido sustituyendo unos a otros.
Este documento ha sido elaborado desde el Laboratorio de Referencia e Investigación de Virus Respiratorios en el Centro Nacional de Microbiología del ISCIII. Sus autores son Sonia Vázquez-Morón, Inmaculada Casas, Vicente Mas, Francisco Pozo, María Iglesias-Caballero y los miembros de RELECOV. Actualmente, el equipo ya está trabajando en el análisis de los últimos meses, como parte del trabajo continuado de vigilancia.
La Red RELECOV está formada por 49 laboratorios distribuidos por todas las comunidades autónomas, que llevan desde 2021 realizando la secuenciación genómica, el estudio y la vigilancia del SARS-CoV-2 en España. Su labor es fundamental, ya que permite obtener un conocimiento indispensable para seguir el comportamiento y evolución del coronavirus, apoyar su estudio epidemiológico y, dado que el virus evoluciona y se adapta, optimizar el desarrollo de vacunas.